2023.07.31
HyenaDNA:
DNAの言語を読み解くLLMの新たなる応用
TL;DR
- 前回のBlogでHyenaというTransformer並の性能を達成しつつ、既存のLLMが抱えていた入力長の二乗で計算コストが増える問題を解決する深層学習モデルを紹介しました。そして、このHyenaを遺伝子解析に応用したHyenaDNAという研究成果が新たに発表されました。
- HyenaDNAでは、ヒトのゲノムをヌクレオチド単位(ATGC)で、次のトークンを予測するタスクで事前学習をします。その後は、HyenaDNAのパラメータは固定し、入力に付加されるプロンプトをファインチューニングすることで、分類問題などの個別のタスクを学習します。
- HyenaDNAは100万トークンもの長いシークエンスの処理に成功しました。これは、既存の研究よりも500倍以上長く、また、HyenaDNAは処理速度も160倍も高速です。そして、HyenaDNAは、各種のベンチマークに対して、既存のモデルの性能を上回りState-of-the-Artを達成しました。
Table of Content
Introduction
こんにちは、グループ研究開発本部・AI研究室のT.I.です。前回のBlogでは、Transformerの弱点である入力長の二乗で計算コストが増える問題を解決するHyenaいう機械学習モデルを紹介しました。これにより今後はLLMが更に長い文章に対応できると期待されます。具体的な長い入力の例として、前々回のBlogでHarry Potterシリーズが200万トークンもあれば余裕で全巻入力できることを取り上げましたが、正直、そんなに長い文章を入力する機会は殆ど無いのでは思っていました。ところが、以外な展開がありまし。なんとHyenaDNAというHyenaをゲノム解析に応用した研究成果が発表されました(Hazy Research による公式の解説Blog HyenaDNA: learning from DNA with 1 Million token context)。このHyenaDNAでは、ヌクレオチド単位の解像度で、なんと100万もの長さのシークエンスの処理に成功し、各種のベンチマークに対してState-of-the-Art(SotA)を達成しました。今回のBlogでは、LLMの新たな応用例としての生命科学へ適用したHyenaDNAという最新の研究成果を紹介します。なお、HyenaDNAのコードや学習で利用したマシンのスペックなどの詳細、モデル・パラメータはHazy Researchの公式GitHubレポジトリ(https://github.com/HazyResearch/hyena-dna)とHugging Face LongSafari (https://huggingface.co/LongSafari)にて公開されており、Google Colabで実行可能です。
HyenaDNA
DNAは、アデニン(A)、チミン(T)、グアニン(G),そして、シトシン(C)の4種類の塩基からなる配列で構成されています。この遺伝情報に基づきタンパク質が合成され、それが我々の生命活動を支えています。驚くべきことに、ヒトの場合、タンパク質をコードする遺伝子は全体の僅か2%程度に過ぎません。残りの98%は、いわゆるジャンクDNA(non-coding region)と呼ばれています。しかし、ジャンクと言っても無駄な領域ではありません。遺伝子の発現は、RNA polymeraseが結合し、mRNAへと転写され開始されます。そのプロセスをコントロールするプロモーター(RNA polymeraseが結合する領域)、サイレンサー(転写活性を弱める領域)、エンハンサー(転写を活性化させる領域)などがジャンクDNAに存在します。特にエンハンサーは、数十万塩基対も離れた位置にある遺伝子の発現をも制御することも知られています。したがって、これらの機能の解明には、非常に長いシークエンスで、長距離離れたような配列間の相互作用の解析が必要です。しかし、これまでに利用できたモデルでは、長大なシークエンスを扱うことが難しいという限界がありました。また、自然言語の場合とは異なり、ヌクレオチド配列の場合、たった一文字(single nucleotide polymorphism (SNPs))の違いが大きな差をもたらす可能性もあります。そのため、ヌクレオチド単位での解像度を持ちながらも長距離のコンテクストを理解する性能が不可欠です。なお、 ヒトのゲノムはおよそ32億の塩基配列からなり、10万以上の長距離の関係性を解明が必要となります。従来のTransformerを利用するfoundation modelでは、 高々512から4,096までのシークエンスしか処理できませんでした(The Nucleotide Transformer)。これはヒトのゲノムの0.001%未満のごく一部の領域に限られます。
このような長距離のコンテクストを理解するために、まさに先日発表されたHyenaは最適です。以下は、シークエンスの長さとPerplexity(予測精度)をTransformerとHyenaDNAで評価した結果です。Transformerでは、4,096までの長さが限界でしたが、HyenaDNAの8層のモデルでは、100万シークエンスまで処理可能です。これは従来の限界の500倍もの長さで、入力長を増やすほど性能(Perplexity)が向上(数値が減少)することが確認できます。
Human Genomeに対するNext token predictionの事前学習の結果。入力長が長くなるほど性能(perplexity)が高まる
では、具体的なモデル構造を見ていきます。HyenaDNAは、order 2のHyena operatorを利用したdecoder-onlyのsequence-to-sequence modelとなります。
入力長\(L\)のベクトル\(x \in \mathbf{R}^L\)に対して、\(x_1, x_2, v \)を以下のHyena projectionで変換します。
$$
x_1 = W_{x_1} u, \qquad x_2 = W_{x_2} u, \qquad v = W_v u
$$
\(W_{x_1}, W_{x_2}, W_v \in \mathbf{R}^{D \times D}\)の行列です。そして、Hyena operatorの出力は以下のようになります。
$$
\begin{eqnarray}
(x_1, x_2, v) &\mapsto& H(x_1, x_2) v \\
H(x_1, x_2) &=& D_{x_2} T_h D_{x_1} \\
\end{eqnarray}
$$
ここで、\(T_h \in \mathbf{R}^{L \times L}\)は、Toeplitz matrix \((T_h)_{ij} = h_{i-j}\)であり、この要素はHyena Filter \(h_t = \gamma_\theta(t)\)のパラメータから生成されます。そして、\(D_{x_1}, D_{x_2} \in \mathbf{R}^{L \times L}\)は、入力に応じて変化するDiagonal matrixです。Hyenaの処理は\(\mathcal{O}(L\log_2 L)\)でスケールするため、Transformerよりも効率的です。HyenaDNAはこれにMLPの層を追加したHyenaDNAブロックを複数積み重ねたものとなります。
入力長\(L\)のベクトル\(x \in \mathbf{R}^L\)に対して、\(x_1, x_2, v \)を以下のHyena projectionで変換します。
$$
x_1 = W_{x_1} u, \qquad x_2 = W_{x_2} u, \qquad v = W_v u
$$
\(W_{x_1}, W_{x_2}, W_v \in \mathbf{R}^{D \times D}\)の行列です。そして、Hyena operatorの出力は以下のようになります。
$$
\begin{eqnarray}
(x_1, x_2, v) &\mapsto& H(x_1, x_2) v \\
H(x_1, x_2) &=& D_{x_2} T_h D_{x_1} \\
\end{eqnarray}
$$
ここで、\(T_h \in \mathbf{R}^{L \times L}\)は、Toeplitz matrix \((T_h)_{ij} = h_{i-j}\)であり、この要素はHyena Filter \(h_t = \gamma_\theta(t)\)のパラメータから生成されます。そして、\(D_{x_1}, D_{x_2} \in \mathbf{R}^{L \times L}\)は、入力に応じて変化するDiagonal matrixです。Hyenaの処理は\(\mathcal{O}(L\log_2 L)\)でスケールするため、Transformerよりも効率的です。HyenaDNAはこれにMLPの層を追加したHyenaDNAブロックを複数積み重ねたものとなります。
HyenaDNAでタスクを解くのは2つのステップに分けられます。最初のステップは、事前学習です。この段階では、次のトークンを予測するタスクが行われます。これは大言語モデル(LLM)と同様のプロセスで、HyenaDNAでは与えられたヌクレオチド配列に基づいて次のヌクレオチドを予測します。HyenaDNAでは“A”, “T”, “C”, “G”の4つのトークンに加えて、その他を示す“N”の5種類と、paddingなどで付加される特殊トークンを用います。事前学習が終了すると、HyenaDNAのパラメータは固定され、次に個別のタスクに対してのファインチューニングを行います。ファインチューニングでは、タスクの入力シークエンスの前にソフト・プロンプト・トークンを付加し、これをHyenaDNAに与えます。プロンプトをチューニングすることで特定のタスクに対して最適化させます。LLMをファインチューニングする際に、このようなプロンプトを介する手法を取ることがありますが、それと同様のプロセスとなります。(例えとして不適切かもしれませんが、“ゲノムの文法”で個別のタスクを解く指示する文章を作成するという感じでしょうか。)以下は、HyenaDNAの事前学習とファインチューニングの概要です。
HyenaDNAの事前学習とファインチューニング(図は論文を元に作成)
では、HyenaDNAの概要を説明したので、具体的な学習データとタスクの解説に進みたいと思います。
HyenaDNAでは、20万以上の長いシークエンスでも安定して訓練するために、初期段階では短いシーケンスから始め、徐々に長いシークエンスの学習へと移行します。このような入力長の変更に柔軟に対応できるのも、Hyenaの畳み込み処理が入力長に応じて動的に変化するHyena Filterを利用していることによります。このWarm-up処理により、45万トークン以上の入力に対して訓練時間は40%短縮され、精度は7.5%ポイント向上しました。Warm-upの概念をアニメーション化したものと、具体的な精度の改善を示したものが以下です。
DNA Sequence length warm-upの概念図(図は論文を元に作成)
Non-warmupとWarmupの訓練時間と精度の関係性。Warmupの方が短時間で効率的に精度が改善されていく。
HyenaDNAの事前学習のデータはHuman Reference Genomeを利用します。HyenaDNAとTransformerの処理時間を比較した結果が以下のずです。短い入力長の場合、Hyenaの諸々のオーバーヘッドのためにTransformerの方が高速です。しかし、長い入力長になるとTransformerの二乗で増える計算コストにより、Hyenaの方が高速になり100万トークンでは、160倍以上の差が生じます。なお、これらのパフォーマンスはGPUはA100 80GBのメモリーで、2層のモデルを用いて比較しています。
TransformerとHyenaDNAを2 layerで比較した場合の処理速度と入力長の関係。100万トークンの場合、HyenaDNAは160倍Transformerよりも高速(A100 80GBを利用)。
次に、GenomicBenchmarksの結果を紹介します。これは8種類の調節因子を予測するデータセットです。(うち7つは2値分類で、1つは3値)入力シークエンスの長さは200-500から最大4,776です。 ベースラインのCNNモデルの結果、そして、Transformerによる結果を抑えてHyenaDNAは全8種のタスクにおいてSotAを達成しています。
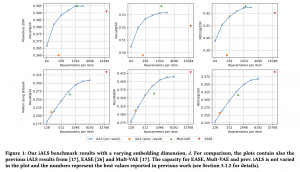
次に、Nucleotide Transformer(NT)との比較結果を紹介します。これらは、200-600塩基長のDNAシークエンスに対して、調節因子を予測するタスクになります。モデル名の後ろの数字は、それぞれモデルのパラメータ数(例500M, 2.5B)と学習に利用したゲノムの数(NTの場合、Human Reference Genome(1)に加えて 世界中の様々な地域のヒト(3202)や他の生物種(850)のゲノムを加えた学習もしています)を示しています。多数のタスクにおいて、HyenaDNAはNTに比べて大幅に少ないパラメータながらも、SotAを達成しております。NTが優れているタスクも存在しますが、これらは多種多様なゲノムを学習データとして用いているのに対し、HyenaDNAはHuman Reference Genomeの1種のみを学習データとして利用しています。これを考慮すると、HyenaDNAの性能は今後さらに向上する可能性があります。
Nucleotide Transformerベンチマークの性能比較(図は論文を元に作成)
次に、HyenaDNAにおける“in-context learning”の性能を見ていきます。ファインチューニングでは、事前に学習したHyenaDNAのモデル・パラメータは固定されています。そこに、入力の前段にソフト・プロンプト・トークンを追加し、このプロンプトを調整することで、特定のタスクに対して最適化します。以下の図は、利用するプロンプトの長さを変更したときの各種ベンチマークタスクの性能を比較したものです。一定の長さ以上のプロンプトを使用することで、モデルの性能が向上することが確認できます。
ソフト・プロンプトの長さと各種ベンチマークの性能比較(図は論文を元に作成)
なお、この論文ではLLMでしばしば利用されるfew-shotプロンプトによる学習も試しました。具体的には、入力 \(X_i \)と正解ラベル\(Y_i\)の組みを以下のように与えます。
$$
\{ X_1, \mathrm{SEP}, Y_1, \mathrm{SEP}, X_2, \mathrm{SEP}, Y_2, \mathrm{SEP}, \dots, X_k, \mathrm{SEP}, Y_k, \mathrm{SEP}, X \}
$$
しかしながら、この\(k\)-shotによる性能改善は、\(k\)を増やしても必ずしも効果がありませんでした。
$$
\{ X_1, \mathrm{SEP}, Y_1, \mathrm{SEP}, X_2, \mathrm{SEP}, Y_2, \mathrm{SEP}, \dots, X_k, \mathrm{SEP}, Y_k, \mathrm{SEP}, X \}
$$
しかしながら、この\(k\)-shotによる性能改善は、\(k\)を増やしても必ずしも効果がありませんでした。
これまでのベンチマークは、比較的短い入力長に対して行われましたが、更に長い入力に対する性能評価をみていきます。DeepSEA data setを利用したChromatin profile predictionのタスクの結果を紹介します。これは、DNAの配列から、転写因子(TF)の結合部位や、DNase(デオキシリボヌクレアーゼ) Iのハイパーセンシティブサイト(DHS)、ヒストンマーク(HM)などの特徴量を予測するタスクです。これらの非コーディング領域の特徴は、遺伝子発現の調節に重要な役割を果たしています。事前学習済みのHyenaDNAをこのタスクにファインチューニングをしたところ、他のベースラインのモデル(DeepSeaはCNN、BigBirdはsparse attentionを使用)よりもすぐれた性能を発揮します。しかも、HyenaDNAではパラメータ数がDeepSea CNNやBigBirdよりもおよそ5倍から30倍少ないです。図中のモデル名の後ろの数字は、それぞれ入力長を表しています。
DeepSEA dataset benchmarkの各タスクのAUROCの比較(図は論文を元に作成)
DeepSEA dataset benchmarkのモデルのパラメータ数の比較(図は論文を元に作成)
最後に、Species Classificationのタスクの結果を紹介します。これは、5種類の動物(ヒト、キツネザル、マウス、ブタ、カバ)のDNAから、その種を予測します。実際のところ、ヒトとその他の哺乳類の遺伝子のほとんどが共通で際は10%未満です。そのため、より正確に判定するには、長いシークエンスを取り扱う必要があります。長い入力に対応できるHyenaDNAでは、シークエンスが長くなるほど精度は改善します。一方でTransformerの場合では、32,000までは改善しますが、それ以上の長さでは学習時間の増大のために適用できません。
Species classificationのタスクの入力長とタスクの性能の比較(図は論文を元に作成)Transformerの場合、25万以上の入力に適用できない。
Summary
今回のBlogでは、前回紹介したHyenaに関する最新の研究成果であるHyenaDNA紹介しました。Hyenaの長い入力長へ対応できるという特徴を活かして、DNAのシークエンスを入力としたタスクにおいて、HyenaDNAは各種のベンチマークでSotAを達成しました。また、HyenaDNAは他のモデルと比較して、モデル・パラメータが少なくて高速という特徴もあります。LLMを利用した新しい分野への挑戦ということで、この種の生命科学への応用は大変に興味深く今後の研究が期待されます。
グループ研究開発本部 AI研究開発室では、データサイエンティスト/機械学習エンジニアを募集しています。ビッグデータの解析業務などAI研究開発室にご興味を持って頂ける方がいらっしゃいましたら、ぜひ募集職種一覧からご応募をお願いします。皆さんのご応募をお待ちしています。
References
- HyenaDNA: Long-Range Genomic Sequence Modeling at Single Nucleotide Resolution, https://arxiv.org/abs/2306.15794
- HyenaDNA: learning from DNA with 1 Million token context (https://hazyresearch.stanford.edu/blog/2023-06-29-hyena-dna)
- AI BUSINESS HyenaDNA: A Large Language Model for the Human Genome (https://aibusiness.com/ml/hyenadna-a-large-language-model-trained-on-human-genome-sequences)
- HazyResearch/hyena-dna (https://github.com/HazyResearch/hyena-dna)
- Hugging Face LongSafari (https://huggingface.co/LongSafari)
- The Nucleotide Transformer: Building and Evaluating Robust Foundation Models for Human Genomics. (https://www.biorxiv.org/content/10.1101/2023.01.11.523679v1 )
- Genome Reference Consortium (https://www.ncbi.nlm.nih.gov/grc)
- Genomic Benchmarks (https://github.com/ML-Bioinfo-CEITEC/genomic_benchmarks)
- DeepSEA training dataset (https://github.com/jakublipinski/build-deepsea-training-dataset)
グループ研究開発本部の最新情報をTwitterで配信中です。ぜひフォローください。
Follow @GMO_RD